008 - 文章阅读笔记:混合密度网络:也许你需要的是一颗骰子 - 知乎 - esang
本文最后更新于:3 个月前
参考文章:
混合密度网络:也许你需要的是一颗骰子 - 知乎 - esang
编辑于 2021-04-16 19:14
ps:本文为依据个人日常阅读习惯,在原文的基础上记录阅读进度、记录个人想法和收获所写,关于原文一切内容的著作权全部归原作者所有。

Kaggle上的”Jane Street Market Predictio“竞赛已经告一段落,部分高位选手的方案也陆续公开,骚操作也是层出不穷。今天介绍一个第二名选手使用的混合密度网络(Mixture Density Networks),原帖在:
Jane Street Market Predictionwww.kaggle.com/c/jane-street-market-prediction/discussion/226837
基于通用近似定理,神经网络可以近似任意函数,但有一个潜在的问题,那就是神经网络拟合的比较好的是单峰分布,对多峰分布的拟合能力堪忧。简单来说,就是当一个x对应一个t值的时候,神经网络表现不错,但是当一个x对应多个t的时候就很拉跨了。
alec:
- dice = 骰子
因为从概率的角度,神经网络是在拟合p(t|x)的分布,但在一些随机性或者波动性比较大的环境中,比如股价未来的走势可能是一个分布,而非一个确定的数值。所以在难以做出精确决策,无法得知骰子究竟掷到几的时候,知道骰子每个面的胜率也是非常有价值的。而混合密度网络就是一个去拟合骰子本身的神经网络。
那么问题来了,让神经网络去拟合一个值很好理解,用均方误差不断优化就行了。但去拟合一个分布,就有两个问题:一是如何让神经网络可以表示一个任意的分布,二是用什么样的损失函数训练这个网络。
alec:
- GMM = 高斯混合模型 = gauss mix model
- M = mid = 混合
第一个问题高斯混合模型(GMM)已经给出了答案:「理论上,多个高斯分布的加权和可以近似任意条件概率分布。」 (这种近似其实是有条件的,但是一般情况下可以无视)
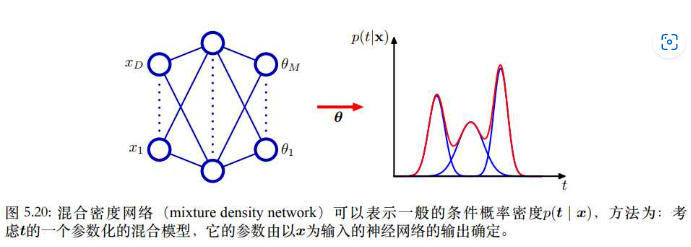
即分布P(t|x)可以表示成K个高斯分布的加权和,第K个高斯分布的权重为πk(x),均值为μk(x), 方差为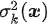 :
:
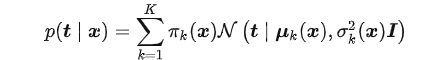
所以我们只需要用三个神经网络分别来拟合这K个分布的权重、均值和方差。当然,因为权重和方差有一些限制:权重要保证和为1,所以使用一个softmax激活函数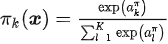 ;方差要保证不为负,所以使用一个指数函数
;方差要保证不为负,所以使用一个指数函数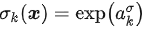
alec:
- 神经网络难以表示非确定性的公式。混合密度网络(MDN),可以用来表示一个条件概率密度。
对于第二个问题,如何设计损失函数。既然我们得到的是一个概率分布,那么按照极大似然估计的原则,这个概率分布应该让我们观测到样本的概率尽可能大。也就是让这个混合概率分布中t的概率尽可能大:
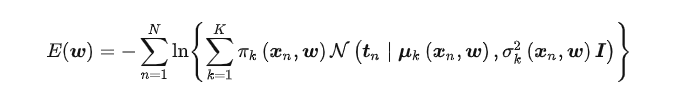
当然,这只是比较原始的混合密度网络,后续出现了一些改进,这里给一个竞赛中的实现:
1 | |